
方显杨研究组与合作者共同开发了一种新型活细胞DNA成像技术
三维基因组互作与表观遗传修饰是基因表达调控的重要因素,其动态变化与细胞生长发育及癌症等疾病的发生发展密切相关。解析染色质在活细胞内的时空动态,是理解基因调控机制的重要科学问题。现有基于CRISPR-Cas系统的成像方法可靶向内源基因序列,但仍受系统复杂性和灵敏度的限制,在非重复序列成像,多位点成像及原代细胞应用方面仍面临挑战。
2025年11月6日,方显杨研究组与合作者在《Nature Biotechnology》期刊在线发表了题为"CRISPR live-cell imaging reveals chromatin dynamics and enhancer interactions at multiple non-repetitive loci"的研究论文。针对前述问题,研究团队将CRISPR技术与拓展遗传密码技术结合,开发出一种新型活细胞DNA成像技术--CRISPR PRO-LiveFISH(Pooled gRNAs with Orthogonal bases LiveFISH)。该技术在此前的CRISPR LiveFISH方法的基础上进行了系统性升级,首次引入扩展遗传字母表中的正交非天然碱基对,实现了对sgRNA的位点特异性荧光标记,并结合对Cas9与sgRNA结构的理性设计,显著提升了探针标记的灵敏度。
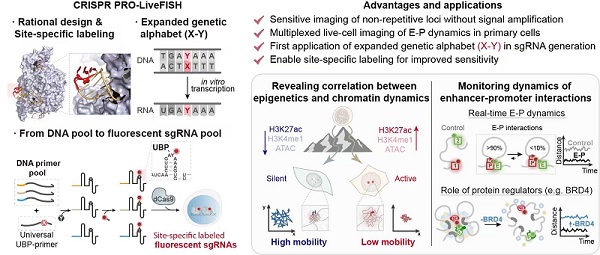
图:CRISPR PRO-LiveFISH 的设计策略及应用
在将LiveFISH技术应用于非重复序列标记时,关键挑战在于如何构建高质量的荧光修饰的gRNA探针库。方显杨研究组此前基于拓展遗传密码系统开发了一系列的RNA定点标记(包括荧光标记)技术(Chem. Sci. 2020, PNAS 2020, Nat. Commun.(2021, 2023, 2025))。PRO-LiveFISH技术通过整合DNA文库与拓展遗传密码技术,采用体外转录与转录后修饰相结合的策略,成功实现了高通量、位点特异性标记的高效sgRNA探针库的制备,进而实现了非重复DNA序列的高灵敏度活细胞成像,并适用于原代细胞。该技术可同时对多达6个不同基因位点进行多色动态观测,并且仅需10条sgRNA即可在活细胞中有效标记非重复序列。在显著简化操作的同时,也可降低对内源基因结构和功能的潜在影响。
利用PRO-LiveFISH技术,研究团队成功实现了活细胞内多基因位点的同步动态观测,揭示了表观修饰和染色质互作相关的动态调控规律。首先,通过比较不同细胞状态(如小分子扰动)与不同细胞类型中同一基因位点的运动行为,发现染色质运动状态与其表观修饰水平密切相关:活跃的组蛋白修饰(如乙酰化)通常伴随着相对受限的位点运动,而乙酰化水平下降位点运动则会相对增强。其次,通过对特定增强子及其靶基因进行同步动态标记,研究发现它们在动态运动中依然能保持持续的空间邻近,提示增强子-启动子互作具有较高的时空稳定性。最后,通过对超级增强子及其靶基因的追踪显示,转录共激活因子BRD4是维持此类三维互作的关键分子,当其功能被抑制时,此类互作便会显著解离。这些发现从动态视角深化了对基因调控机制的理解。
综上,通过结合扩展遗传密码技术与sgRNA理性设计,CRISPR PRO-LiveFISH技术为动态三维基因组研究提供了新的活细胞多色成像工具,适用于非重复DNA序列的动态标记以及原代细胞成像。借助该工具,研究团队系统揭示了染色质动态与表观遗传之间的关联规律、解析了增强子-启动子(E-P)的时空互作特性,并阐释了转录共激活因子BRD4在维持超级增强子三维互作中的关键作用,从而深化了对表观调控和三维基因组时空动态调控机制的理解。
清华大学生命科学学院王海峰研究员、中国科学院生物物理研究所方显杨研究员和清华大学生命科学学院颉伟教授是该论文的共同通讯作者。清华大学博士生刘美铄、黄可韵和已毕业博士生张杰(现为生物物理所方显杨组博士后)为论文共同第一作者。该研究受到国家重点研发计划、国家自然科学基金、中国科学院战略性先导科技专项(B类)和北京市自然科学基金的支持。
文章链接:https://www.nature.com/articles/s41587-025-02887-3
(供稿:方显杨研究组)
附件下载: